Gel-basierte Analytik von
intakten Proteinen
Unsere Hochleistungs-Analytik von intakten Proteinen basiert auf der zweidimensionalen Gelelektrophorese (2DE). Die Proteine werden in zwei Dimensionen aufgetrennt, erstens nach dem isoelektrischen Punkt (pI) und zweitens nach ihrem Molekulargewicht. Dies resultiert in einer äußerst hohen Trennungskapazität von bis zu 2000 Proteinen in komplexen Proteomen. Im Gegensatz zu MS-basierten Analysen bleiben die Proteine während der 2DE-Analyse intakt und werden nicht zu Peptiden verdaut. Somit tragen die Proteine in der 2DE noch alle Informationen wie:
- • Post-translationale Modifikationen wie Phosphorylierungen, Glykosylierungen,
Oxidationen
- • Protein-Isoformen
- • Protein Splicing-Varianten
- • pI-Wert
- • Molekulargewicht
Wir nutzen dabei unsere eigenen Fluoreszenz-Labeling-Technologien und kombinieren dieses mit über 10 Jahren hands-on Erfahrung. Damit können wir Ihnen einen 2DE-Service anbieten, der in punkto Leistungsfähigkeit, Genauigkeit und schneller Verfügbarkeit der Daten weltweit seines Gleichen sucht.
Je nach individueller Fragestellung und Beschaffenheit Ihrer Proben (z.B. Probenmenge, Konzentration, Pufferzusammensetzung) haben wir ein großes Portfolio an Technologien zur Auswahl.
.
2DE Anwendungen und die Analyse von intakten Proteinen (Auswahl)
- • Charakterisierung eines Proteoms (Protein-Muster, PTM- und Proteinisoform-
Verteilung): T-Rex 2D
- • Proteom-Profiling (differentieller Vergleich mehrerer Proteome): Refraction-2D,
Saturn-2D
- • Redox-Status-Analysen: Saturn-2D REDOX
- • Untersuchungen von post-translationalen Modifikationen (Phosphorylierungen
und Glykosylierungen): T-Rex 2D-PHOS und T-Rex 2D-GLYCO
- • 2D Western Blot Analysen: T-Rex 2D WB
- • Host cell protein analysis: T-Rex 2D HCP
.
.

Refraction-2D (DIGE)
Weltweit bekannt als 2D Differential Gel Electrophoresis (2D DIGE) basiert unsere Refraction-2D Technologie auf der Markierung der Proteinproben mit verschiedenen Fluoreszenzfarbstoffen (z.B. G-Dye200 + G-Dye300) und der Verwendung eines internen Standards, der eine Mischung aus allen Proteinproben enthält (G-Dye100).
Zwei markierte Proben und ein interner Standard können gleichzeitig in einem Gel aufgetrennt und analysiert werden. Durch den internen Standard können mehrere Gele miteinander in Beziehung gesetzt und somit direkt verglichen werden. Dies ermöglicht eine sehr genaue Quantifizierung und Identifikation von differentiell exprimierten oder modifizierten Proteinen.
.
Saturn-2D
Wenn Sie nur sehr wenig Material zu Verfügung haben, dann ist oftmals die Saturn-2D Technologie die Methode der Wahl. Sie benötigt, anders als Refraction-2D, nur ein Zehntel der Probenmenge. Aber auch wenn Ihre Proben z.B. stark salzhaltig sind und eine Umpufferung zu viel an Substanzverlust bedeuten würde, kommt Saturn-2D ins Spiel.
.
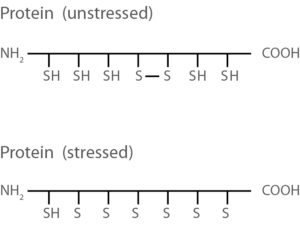
Saturn-2D REDOX
Unsere Saturn-2D REDOX Technologie wurde speziell für die Visualisierung der komplexen Antwort im Proteom von Zellen auf Stress entwickelt. Mit Saturn-2D™ REDOX werden Probenspezies mit unterschiedlichem Redox-Potential spezifisch markiert und nachfolgend miteinander verglichen.
.
T-Rex 2D-Phos/ 2D-Glyc/ 2D-WB/ HCP
Phosphorylierungssanalytik
In Kombination mit einer phosphorylierungsspezifischen Färbung ist der T-Rex die ideale Möglichkeit das Gesamtprotein zu markieren und als interner Standard so eine differentielle Analytik zu ermöglichen. Somit können gesamte Proteine direkt mit den phosphorylierten Proteinen verglichen werden. Im Gegensatz zu Dopplefärbungen (zuerst Gesamtprotein, anschließend Phosphoprotein) wird beim T-REX-Ansatz das Gel geschont und unspezifische Auswaschungen von interessanten Proteinen verhindert.
Glykolysierungsanalytik
In Kombination mit einer glykolysierungsspezifischen Färbung ist der T-Rex die ideale Möglichkeit, das Gesamtprotein zu markieren und als interner Standard so eine differentielle Analytik zu ermöglichen. Somit können gesamte Proteine direkt mit den glykosylierten Proteinen vergleichen werden. Im Gegensatz zu Dopplefärbungen (zuerst Gesamtprotein, anschließend Phosphoprotein) wird beim T-REX-Ansatz das Gel geschont und unspezifische Auswaschungen von interessanten Proteinen verhindert.
2D Western Blotting
T-Rex markierte Proteine lassen sich hervorragend von einem Gel auf eine Blottingmembran (0.22 oder 0.45 µm, NC oder PVDF) transferieren. Damit ist das Protein durch Fluoreszenz-Imaging zu jedem Zeitpunkt des Western Blots detektierbar (z.B. nach Transfer, nach Target-Defektion). Für Western Blots kann der Nachweis des Targets dann mittels Chemilumineszenz, Immuno Blue Fluorescent Substrate oder mit Fluoreszenz-konjugierten Antikörpern erfolgen. T-Rex beeinflusst die Bindung durch Antikörper nicht.
Host Cell Protein (HCP) Analytik
Für die sichere Unterscheidung von wirtseigenen (host cell) und im Wirt rekombinant hergestellten Proteinen ist der 2D Western Blot der Goldstandard. Dabei eignet sich die Markierung des Gesamtproteins mit dem rot-fluoreszierenden T-Rex ideal, um dies nach Transfer auf der Blottingmembran in höchster Sensitivität und binnen Sekunden nachzuweisen. In Kombination mit einem Anti-HCP Antikörper (Fluoreszenz-markiert oder HRP-markiert + ECL) kann dann in einem einfachen, schnellen Overlay die HCP Kontamination sicher bestimmt werden. Diese Methode eignet sich ebenfalls zur optimalen Auswahl des geeigneten anti-HCP Antikörpers.
.
.

Weitere Informationen/ Preisanfragen
Für Fragen, Informationen und Preisangebote setzen Sie sich bitte mit unseren Service-Team in Verbindung, wir beraten Sie gerne:
Mo - Fr 9 -17h
+ 49 (0) 345 2799 6413
service dyeagnostics.com
dyeagnostics.com
.
.

Unsere Produkte
Probenmarkierung
1D Fluoreszenz-Labeling Kits zur
Normalisierung von Western Blots
.
Fluoreszierende Standards
MW Größenstandard (Cal A)
Smartalyzer S + L
.
1D Fluoreszenz-Labeling Kits
für die Gelanalytik
SPL Smart Protein Layers
.
2D Fluoreszenz Labeling Kits
Refraction-2D
Elektrophorese
Fluoreszierende Proteingrößenstandards
.
Fertiggele
VELUM 1D SDS
VELUM 1D Native
.
Elektrophorese
ORCA Gel Electrophoresis research
ORCA Gel Electrophoresis routine
Zusatzelektroden
Gel Frames
Western Blotting
Blotter
BEO Dry Blotter
Trennfolien
Blottingmembranen
Low fluorescent Nitrocellulose Membranes
Low fluorescent PVDF Membranes
Development folders
Immuodetektion
Imaging Systeme
Imager
ORCA Fluorescence Imager
.
Imaging Zubehör
Imaging Tray customized
.
Komplettsysteme
Datenanalyse
Software für 1D Gele & Blots
Software für 2D Gele & Blots
EPO Doping Analysis
Komplettsysteme
ORCA EPO Analyzer
.
Proteingele für EPO-Screenning
Hochdurchsatz SARCOSYL-PAGE:
VELUM Precast SAR Gels
.
Hochdurchsatz IEF:
VELUM Precast IEF Gels
.
Hochdurchsatz SDS-PAGE:
VELUM Precast SDS Gels
.
Fluoreszierende EPO Standards
SEPO
.
Blotting und Blot-Prozessierung
Hochdurchsatz Blotting Kits
.
Hochdurchsatz Washing Tray für BlotCylcler
Two-dimensional gel electrophoresis
The principle of 2DE
The two-dimensional gel electrophoresis (2-D electrophoresis, 2-DE) is the most powerful method to separate complex protein mixtures.
It is based on the separation of intact proteins (not peptides) according to two molecular properties: the electric charge and the size of a protein.
The 2-D electrophoresis starts with the first-dimension separation - proteins are separated according to their electric charge, that is their isoelectric point. This step is called isoelectric focusing (IEF).
The IEF is (now a days) performed on a IEF strip, an acrylamide strip containing a pH gradient. When an electric potential is given to the IEF strip, the proteins will move along the strip to that point where their electric charge is 0 (neutral charge). After the IEF, the IEF strip now holds “a line of” the separated proteins according to their electrical charge.
After a step of equilibration the IEF strip is then applied to a SDS-PAGE gel in order to run the second dimension. This separation step now separates the now denatured proteins by their molecular weight.
.
2DE separation capacity
The protein separation capacity is given by
- a) the capacity of protein load of the IEF strip
- b) the separation area of the second dimension (the size of the SDS-PAGE gel).
For protein extracts from E. coli one should separate about 500 different proteins (e.g. detected by T-Rex fluorescent protein label) with 7 cm IEF strips and mini gel size SDS-PAGE (8 x 10 cm) and about 1200 (e.g. detected by T-Rex fluorescent protein label) proteins when using 24 cm strips and maxi size SDS-PAGE (27 x 22 cm).
.
Protein detection/ visualization
The number of proteins which can be detected is dependent on the method of protein visualization.
The general sensitivity limit is...
- • for Coomassie stains: a protein with an abundance of in the range of 100-10 ng can be detected.
- • for silver nitrate stains: a protein with an abundance of about 10-1 ng can be detected.
- • for fluorescence protein labels (minimal lysine labeling): proteins with an abundance of about 1 – 0.1 ng can be detected*.
- • for fluorescence protein labels (saturation cysteine labeling): proteins with an abundance of about 0.1 – 0.01 ng can be detected*.
* adequate fluorescence detection imaging device required
.
Differential Gel Electrophoresis/ Sample multiplexing
The usage of fluorescent protein labels is a strong benefit of 2D gel electrophoresis as the detection limit and dynamic range of the signal is much better and the analysis does not suffer from protein staining artifacts like background issues, difference in results due to room temperature, freshness of stains, etc… .
However, as proteins carrying a covalently bound fluorescence tag (label), they can be specifically detected. This allows to combine many samples to one 2DE analysis to allow for a precise quantification of protein expression and/or the accurate detection of post-translational protein modifications and to dramatically reduce the number of required 2D gels.
.
Post-translational modifications
| Post-translational modification | Phosphorylation | Glycosylation | RedOx |
| PTM causes
differences in... |
pI (isoelectric point) |
molecular weight and or pI
|
molecular weight and or pI
|
| Detection by... |
|
|
|
| PTM for e.g. |
activation/ deactivation of kinases signal transduction of tumors, signal transduction upon pathogen reception
|
metabolism of tumors
important role in Alzheimers desease, diabetis, ateriosclerosis
|
activation/ deactivation of proteins tumor metabolism apoptosis ROS
|
| PubMed Publications 2016 | 13,716 | 2,300 | 1,149 |
| Pubmed Publications until May 2017 | 6,834 | 1,150 | 543 |
.
Unser Leistungsspektrum
- • Proteomstudien (Shot Gun)
- • Proteomstudien 2DE-basiert
- • Label und labelfreie Proteinquantifizierung
- • Proteincharakterisierung (Quantifizierung, Reinheit, Modifikation, Identifikation)
- • Mutagenisis Discovery
- • Mircoarray Follow-up
- • Drug Target Discovery
- • Assay Development
- • Protein Pattern Analysis
- • toxikologische Fingerprint-Analysen
- • Saatguttestung
- • Proteinverunreinigungen
- • Beratung für komplexe Proteinanalysen
- • Differentielle Proteingelektrophorese (2D-DIGE)
- • Post-translationale Proteinmodifikationen (Phosphorylierungen, Glykosylierungen, Oxidation/ Reduktion)
- • Identifikationen von Isoformen und Splicing-Varianten
- • Biomarker-Discovery
- • 2D Western Blotting
- • HCP Coverage 2D Western Blotting
- • Standardisierte und quantitative SDS- und SAR-PAGE, IEF sowie Western Blots mit bis zu 52 Proben pro Lauf
.
Ausstattung für die Gel-basierte Protein Analytik
- • OCRA Gel Electrophoresis Units
- • Dolphin Gel Electrophoresis Units
- • HOEFER SE900 Gel Electrophoresis
- • HOEFER Gel Electrophoresis small
- • BEO Dry Blotter
- • VELUM Dry Blotter
- • OCTOPLUS QPLEX Fluorescence Imager
- • Typhoon FLA 9000 Imager
- • HOEFER IEF 100
- • LabImage L360 Software
- • Delta2D Software
- • Spot Picker
- • Spot Processing
- • Fluorescent Label
- • VIS- und Fluoreszenz-Stains
.
Ausstattung & Methodik für die Massenspektrometrie
- • Synapt High Definition MS-Systeme (HD-MS, Waters)
- • Q Exactive Plus Hybrid Quadrupole-Orbitrap MS (Thermo)
- • Xevod TQD Tandem Quadrupol MS-System (Waters)
- • MALDI-TOF/TOF (ultraflex, Bruker Daltonics)
- • MS/MS und HD-MSE
- • ESI, nano-ESI, MALDI
- • SRM, PRM und MRM
- • UPLC, nano-UPLC
- • diverse Bioinformatische Tools
















